3 Grafiken erstellen
3.1 Aller Anfang…
… ist eigentlich nicht schwer. Bevor es losgeht müssen wir immer die notwendigen Pakete laden. Da in diesem Kapitel mit ggplot2 gearbeitet wird, muss zumindest das geladen sein. Der Datensatz penguins, den wir uns anschauen, ist ab R 4.5.0 immer als Datensatz geladen, allerdings sind die Merkmalsnamen anders. Sollte die installierte R Version älter sein, so muss das Paket palmerpenguins auf jeden Fall geladen werden. Im Paket ggthemes gibt es Farbpaletten, die auch von Farbenblinden gut auseinander gehalten werden können, weswegen wir auch diese beiden Pakete (installieren und) laden.
Später, wenn Daten nicht in einem Paket gegeben sind, sondern zusätzlich geladen und aufgearbeitet werden müssen, ist es einfacher das komplette Tidyverse (und ggf. andere benutzte Pakete) am Anfang (eines Skriptes) zu laden, da nicht nur ggplot2 sondern auch Pakete wir readr, tidyr, dplyr etc. benötigt werden, die alle Teil des Tidyverses sind. Wir werden in Kapitel 4 noch näher darauf eingehen.
3.2 Erstellen von Grafiken
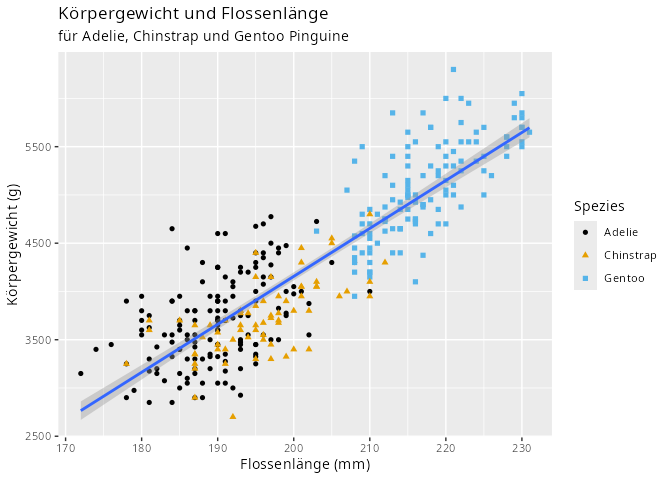
Ziel dieses Kapitels ist es den Aufbau einer ggplot2-Grafik zu verstehen, und am Ende der Kapitels wollen wir aus der Datentabelle penguins die folgende Grafik erstellen können:
3.2.1 Die Datentabelle penguins
Nachdem wir das Paket palmerpenguins geladen haben, steht der Datensatz penguins zu Verfügung, den wir nutzen, um die Funktionsweise von ggplot2 zu verstehen. Durch Eingabe von penguins in der Konsole können wir uns den Datensatz (bzw. die ersten Zeilen des Datensatzes) ansehen.
penguins# A tibble: 344 × 8
species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g
<fct> <fct> <dbl> <dbl> <int> <int>
1 Adelie Torgersen 39.1 18.7 181 3750
2 Adelie Torgersen 39.5 17.4 186 3800
3 Adelie Torgersen 40.3 18 195 3250
4 Adelie Torgersen NA NA NA NA
5 Adelie Torgersen 36.7 19.3 193 3450
6 Adelie Torgersen 39.3 20.6 190 3650
7 Adelie Torgersen 38.9 17.8 181 3625
8 Adelie Torgersen 39.2 19.6 195 4675
9 Adelie Torgersen 34.1 18.1 193 3475
10 Adelie Torgersen 42 20.2 190 4250
# ℹ 334 more rows
# ℹ 2 more variables: sex <fct>, year <int>Der Datensatz enthält 8 Merkmale (die Spalten) und 344 Beobachtungen (die Zeilen). Die Tabelle zeigt eine Erläuterung der Merkmale, die man auch in der Hilfe help(penguins) findet.
penguins aus dem Paket palmerpenguins
| Name des Merkmals | Bedeutung | Skalentyp | in R |
|---|---|---|---|
species |
Pinguin Spezies | nominal | <fct> |
island |
Insel im Palmer-Archipel | nominal | <fct> |
bill_length_mm |
Schnabellänge in mm | metrisch | <dbl> |
bill_depth_mm |
Schnabeltiefe in mm | metrisch | <dbl> |
flipper_length_mm |
Flossenlänge in mm | metrisch | <dbl> |
body_mass_g |
Körpergewicht in g | metrisch | <int> |
sex |
Geschlecht | nominal | <fct> |
year |
Jahr der Untersuchung | metrisch | <int> |
Beim Aufrufen der Hilfefunktion in der Konsole mittels
help(penguins)
# oder alternativ
?penguinserscheint die Hilfe im dafür vorgesehenen Reiter. Die geschweifte Klammer in der Hilfe penguins {palmerpenguins} bedeutet, dass der Datensatz (allgemein das R-Objekt) im Paket palmerpenguins enthalten ist. Einige Pakete wie zum Beispiel base, datasets, utils oder stats sind immer geladen, wenn R geöffnet wird.
penguins
Eine weitere Möglichkeit eine Übersicht über die Daten zu bekommen, ist die Funktion glimpse(), die im Paket dplyr aus dem Tidyverse enthalten ist.
glimpse(penguins)Rows: 344
Columns: 8
$ species <fct> Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adel…
$ island <fct> Torgersen, Torgersen, Torgersen, Torgersen, Torgerse…
$ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3, 38.9, 39.2, 34.1, …
$ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6, 17.8, 19.6, 18.1, …
$ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181, 195, 193, 190, 186…
$ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650, 3625, 4675, 3475, …
$ sex <fct> male, female, female, NA, female, male, female, male…
$ year <int> 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007…In der ersten Spalte stehen die Merkmale des Datensatzes, dahinter der Datentyp und in der dritten Spalte die ersten Einträge des jeweiligen Merkmals.
3.2.2 Erste Schritte zur Grafik
Um Daten zu untersuchen sind geeigneten Grafiken ein sehr gutes Mittel. Welche Grafiken geeignet sind hängt von der Anzahl der Merkmale, die dargestellt werden sollen, sowie deren Skalenniveaus ab. Darauf werden wir im Kapitel 16 und im Kapitel 14 noch genauer eingehen. Neben dem explorativen Charakter haben Grafiken in wissenschaftlichen Publikationen, Bachelor-, Master- oder Doktorarbeiten die Aufgabe Informationen möglichst verständlich zu übermitteln.
Wir fangen mit einem einfachen Streudiagramm (zwei metrische Merkmale) an, und untersuchen den Datensatz penguins auf die Frage
“Gibt es einen Zusammenhang zwischen Flossenlänge und Körpergewicht bei Pinguinen?”
Die Merkmale des Datensatzes um dieser Frage nachzugehen sind flipper_length_mm und body_mass_g. Wie wollen nun die Grafik Schritt für Schritt aufbauen. Dabei erkennt man sehr schnell, dass der Aufbau in Lagen (auf englisch: layers) funktioniert.
Die Funktion ggplot() stellt das Grundgerüst jeder Grafik dar.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
)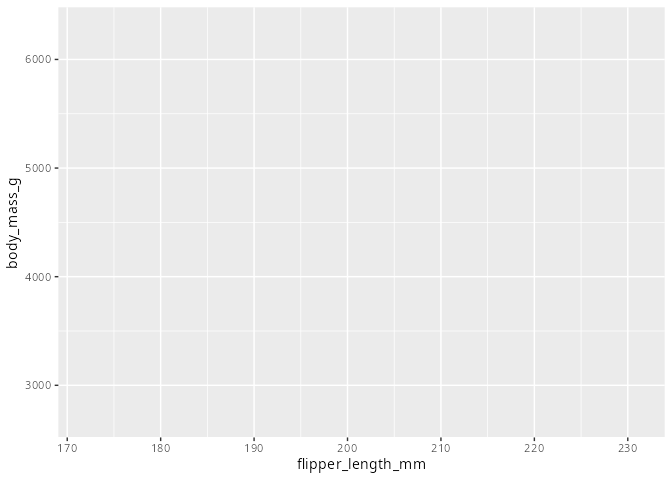
Gehen wir jeden Schritt oben einmal durch: Dem Argument data= wird die Datentabelle (hier: penguins) zugewiesen. Im Argument mapping= wird angegeben, wie die Merkmale des Datensatzes zu verwenden sind. Dies geschieht innerhalb der Funktion aes(), was für Aesthetics steht. In unserem Beispiel wird der x-Koordinate das Merkmal flipper_length_mm zugewiesen und der y-Koordinate das Merkmal body_mass_g. Damit hat man nun ein leeres Koordinatensystem geschaffen wie Abbildung 3.3 zeigt. Der dargestellte Abschnitt der Achsen wird so gewählt, dass alle Datenpunkte dargestellt werden könnten. Wir werden später noch lernen, wie man die dargestellten Achsenabschnitte ändern kann.
Im nächsten Schritt wird der Grafik die geometrische Funktion geom_point() hinzugefügt. Dies geschieht mit einem + Zeichen hinter der Funktion ggplot(). Die Funktion geom_point() zeichnet nun das Streudiagramm, das heißt für jede Beobachtung wird der flipper_length_mm / body_mass_g in das Koordinatensystem eingezeichnet.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
) +
geom_point()Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).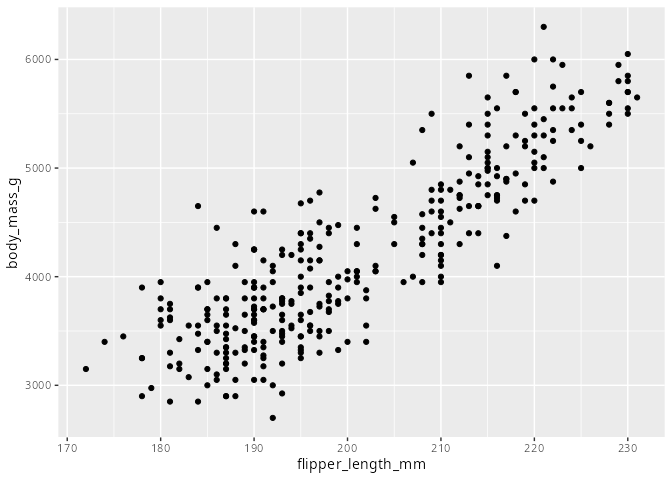
Dieses Streudiagramm ist noch relativ weit weg von Abbildung 3.1, allerdings kann man an dieser Stelle bereits die Frage “Gibt es einen Zusammenhang zwischen Flossenlänge und Körpergewicht bei Pinguinen?” beantworten, da der Trend “von unten links nach oben rechts” der Punkte im Koordinatensystem gut zu sehen ist.
Bemerkung:
Zwischen dem R-Code und der Grafik steht die Warnung:
| Warning: Removed 2 rows containing missing values or values
Dies bedeutet, dass bei zwei Beobachtungen mindestens eines der beiden Merkmale ein Wert fehlt. An Stelle eines Wertes steht NA, was für Not Available steht, in der Datentabelle. Daher kann für diese Beobachtung kein Punkt in der Grafik dargestellt werden.
Dies ist ein typisches Verhalten für R (sofern man bei Programmiersprachen von einem Verhalten reden kann): es werden keine Daten einfach unterschlagen, sondern der Benutzer soll wissen wenn R Daten nicht oder nur partiell darstellen kann. Es gibt einige Stellen an denen R den Nutzer warnt, da dieser entweder ungünstige Entscheidungen getroffen hat oder ggf. für den Nutzer unerwartete Ausgaben produziert wurden.
3.2.3 Hinzufügen von Aesthetics und Schichten
Der nächste Schritt beim Aufbau der Grafik ist nun zusätzlich zu den zwei metrischen Merkmalen ein kategoriales Merkmal, nämlich die Spezies, die im Merkmal species steht, hinzuzufügen. Wir möchten, die Punkte je nach Spezies verschieden einfärben. Das Argument dazu heißt color= oder colour= und muss ebenfalls in die Funktion aes() geschrieben werden. Der Grund ist, dass alle Eigenschaften der Grafik, die von der Ausprägung eines Merkmals abhängig sind, innerhalb der Funktion aes() stehen müssen.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g, color = species)
) +
geom_point()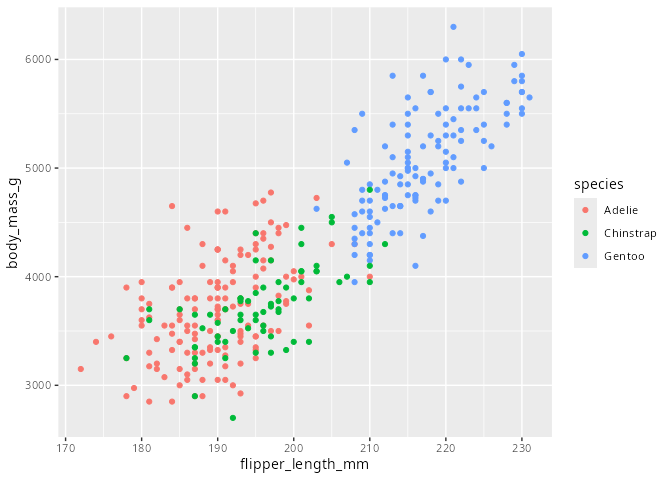
Der nächste Schritt besteht darin eine Regressionsgerade durch die Daten zu legen. Die erreichen wir durch Hinzufügen einer weiteren geometrischen Funktion geom_smooth(). Ohne weitere Argumente hinzufügen wird eine geglättete Kurve mit Konfindezintervall eingezeichnet.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g, color = species)
) +
geom_point() +
geom_smooth(method = "lm")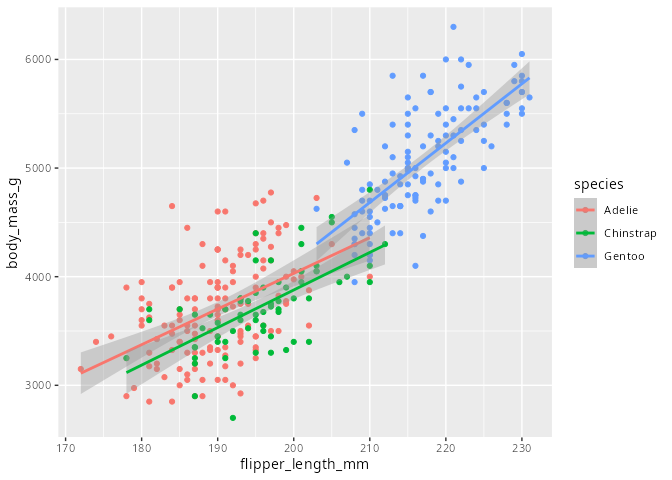
Das Argument method= "lm" sorgt dafür, dass die geglättete Kurve mit Hilfe einer linearen Regression gewonnen wird. Wie man erkennen kann, werden in obigen Fall die Regressionsgeraden für jede Spezies einzeln ermittelt. Das ist allerdings nicht das, was wir im Sinn hatten. Beheben kann man dies indem die Aesthetic für die Einfärbung color= nur für die geometrische Funktion geom_point(), nicht aber für alle anderen geometrischen Funktionen verwendet werden soll.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
) +
geom_point(mapping = aes(color = species)) +
geom_smooth(method = "lm")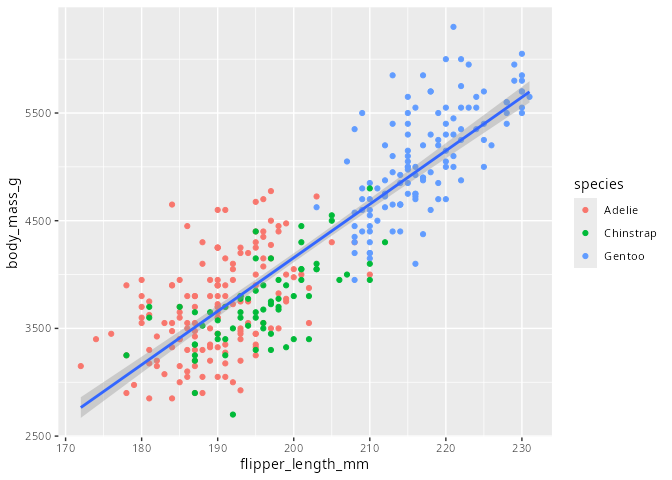
Wir sehen, dass mapping= in der Funktion ggplot() global für alle geometrischen Funktionen gilt, man jedoch auch lokal Aesthetics für einzelne geometrische Funktionen angeben kann.
Das Ergebnis ist schon sehr nah an Abbildung 3.3. Da innerhalb des Streudiagramms nicht nur die Farbe, sondern auch die Form der Datenpunkte bezüglich des Merkmals species geändert ist, kann man dies noch ändern. Dafür wird das Argument shape= in die Aesthetics geschrieben. Da für jede Spezies die Datenpunkte im Koordinatensystem eine andere Form bekommen sollen, lautet das Argument shape=species.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
) +
geom_point(mapping = aes(color = species, shape = species)) +
geom_smooth(method = "lm")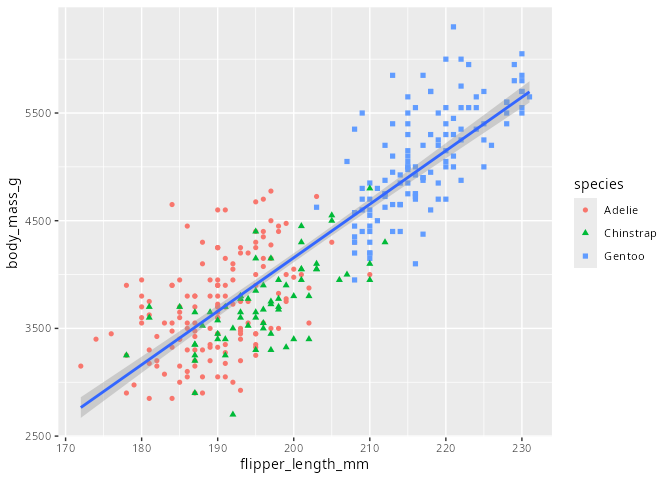
Die beiden letzten Änderungen betreffen die Beschriftungen, sowie das Ändern des Farbschemas.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
) +
geom_point(mapping = aes(color = species, shape = species)) +
geom_smooth(method = "lm") +
labs(title = "Körpergewicht und Flossenlänge",
subtitle = "für Adelie, Chinstrap und Gentoo Pinguine",
x = "Flossenlänge (mm)",
y = "Körpergewicht (g)",
color = "Spezies",
shape = "Spezies") +
scale_color_colorblind()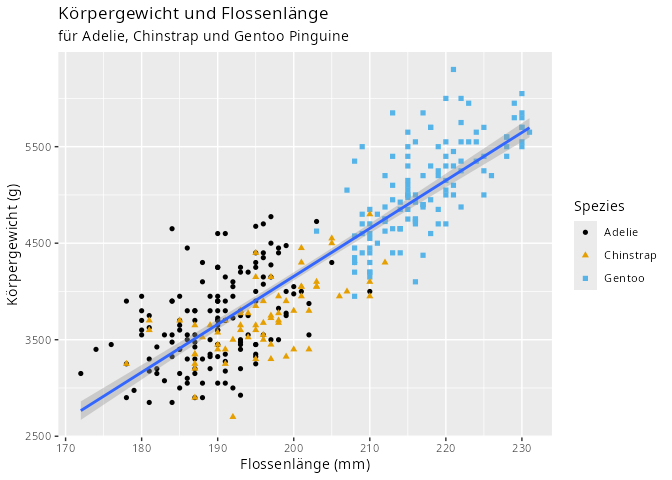
Für die Änderungen aller Beschriftung fügt man als nächste Schicht die Funktion labs() hinzu und gibt an, welche Größen umbenannt werden sollen. Interessant sind die letzten beide Argumente: color= und shape= Damit wird in der Legende die Überschrift geändert.
Die letzte Funktion scale_color_colorblind() aus dem Paket ggthemes ändert das Farbschema derart, dass es für Farbenblinde geeignet ist.
3.2.4 Die ggplot2-Syntax
Der Code der obigen Grafiken war sehr explizit und etwas sperrig. Um die Syntax ein wenig zu vereinfachen, schauen wir uns den Code der Grafiken noch einmal an und vereinfachen die Syntax.
ggplot(
data = penguins,
mapping = aes(x = flipper_length_mm, y = body_mass_g)
) +
geom_point(mapping = aes(color = species))Sehen wir uns dafür einmal die Hilfe der Funktion ggplot() an:
ggplot()
Die ersten beiden Argumente der Funktion ggplot() sind data= und mapping= und bei den geometrischen Funktionen wie geom_point() ist mapping= das erste Argument. Bei Funktionen, die man häufiger nutzt, lässt man den Namen des ersten, ggf. auch der ersten beiden Argumente in der Regel weg. Dies erleichtert die Lesbarkeit des Codes. An den allermeisten Stellen in diesem Kurs werden wir auch auf diese Argumentnamen verzichten. Der obige Code wird damit zu:
ggplot(penguins, aes(x = flipper_length_mm, y = body_mass_g)) +
geom_point(aes(color = species))Nutzen wir nun noch die Pipe-Syntax (siehe Kapitel 5.5), so erhalten wir eine kompakte und praktische Form um Grafiken zu erzeugen. Dass diese Schreibweise sehr vorteilhaft ist wird spätetstens im Kapitel 8 klar.
penguins |>
ggplot(aes(x = flipper_length_mm, y = body_mass_g)) +
geom_point(aes(color = species))Bemerkungen
Sowohl der Pipe-Operator
|>als auch das Plus-Zeichen+dürfen nur in der der Mitte oder am Ende einer Zeile, nicht aber am Anfang einer Zeile stehen. R interpretiert eine Zeile in die nicht mit einer Pipe oder einem Plus endet als vollständig (sofern alle Klammern geschlossen wurden), und führt diese dann nur bis dahin aus!Bei R sind die Einrückungen der Zeilen wie im obigen Code nicht notwendig. Bei andern Programmierspechen, wie zum Beispiel Python, ist das anders, da dort keine Klammern gesetzt werden. Ein Einrücken der Zeilen, sowie gezielt gesetzte Leerzeilen erhöht aber die Lesbarkeit des Codes. Es gibt einen Tidyverse Style Guide in dem erläutert wird wie man Code setzen soll. Dies ist insbesondere dann interessant, wenn man ein Skript schreibt und nicht nur Code in die Konsole eintippt.
3.3 Grafiken abspeichern
Nachdem man eine Grafik erstellt hat, möchte man diese speichern, um sie irgendwoanders zu nutzen. Dies geht mit der Funktion ggsave(), die die erzeugte Grafik in das Arbeitsverzeichnis abspeichert:
penguins |>
ggplot(aes(x = flipper_length_mm, y = body_mass_g)) +
geom_point()
ggsave(filename = "pinguin-plot.png")Es ist auch möglich der Funktion die Argumente width= und height= mitzugeben, so dass man die Größe der Grafik kontrollieren kann. Gibt man die Argumente nicht an, so werden die Größen vom aktuellen plotting device genommen.
Viel besser als die Grafiken abzuspeichern und irgendwo einzufügen ist allerdings die Verwendung von quarto, da man dort die Grafiken direkt dynamisch einbinden kann.