# A tibble: 6 × 4
country year cases population
<chr> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 12804285839 Datentabellen bereinigen
Fangen wir das Kapitel doch gleich mit einem kleinen Quiz an. Gegeben sind vier verschiedene Darstellungen (1), (2), (3) und (4) von einem Datensatz. Welche der Darstellungen ist vermutlich am einfachsten zu benutzen?
# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583# A tibble: 6 × 3
country year rate
<chr> <dbl> <chr>
1 Afghanistan 1999 745/19987071
2 Afghanistan 2000 2666/20595360
3 Brazil 1999 37737/172006362
4 Brazil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583# A tibble: 3 × 3
country `1999` `2000`
<chr> <dbl> <dbl>
1 Afghanistan 745 2666
2 Brazil 37737 80488
3 China 212258 213766# A tibble: 3 × 3
country `1999` `2000`
<chr> <dbl> <dbl>
1 Afghanistan 19987071 20595360
2 Brazil 172006362 174504898
3 China 1272915272 1280428583Im Laufe des nächsten Kapitels möchten wir die Vor- und Nachteile der verschiedenen Darstellungen diskutieren. Wir werden die Funktionen mit denen man unliebsame Datentabellen aufräumen und in eine geeignete, gut zu verwendende Darstellung bringen kann.
9.1 Bereinigte Daten
In diesem Kapitel geht es darum, Datensätze zu bereinigen (tidy data). Dazu wird zusätzlich zu dplyr das Paket tidyr aus dem Tidyverse benötigt. Aber was genau sind bereinigte Daten und wieso ist das ein Thema mit dem man sich auseinandersetzen muss? Die Datensätze mit denen wir bisher gearbeitet haben (penguins aus palmerpenguins, diamonds, flights aus dem Paket nycflights13 etc.) waren immer bereinigt, das heißt
- jede Spalte entspricht genau einem Merkmal,
- jede Zeile enthält genau eine Beobachtung und
- in jeder Zelle steht genau ein Wert.
Die drei Bedingungen hängen offenbar voneinander ab, und es ist nicht möglich, nur zwei der drei Regeln zu befolgen. Dieser Zusammenhang erlaubt es, einen noch einfacheren Satz von praktischen Anweisungen zu formulieren:
- Stelle die Daten in einer Datentabelle (einem Tibble) dar.
- Definiere die Spalten so, dass sie genau ein Merkmal enthalten.
9.2 Warum sollte man die Daten bereinigen?
Es ist vernünftig Daten auf eine in sich konsistente Art und Weise aufzubewahren. Dadurch wird es wesentlich einfacher, mit bekannten Werkzeugen zu arbeiten und diese immer wieder auf ähnliche Art und Weise anzuwenden.
Vektoren sind die grundlegende Struktur in R. Merkmale in Spalten anzuordnen ermöglicht es, diese Struktur in vollem Maße zu nutzen. Funktionen wie
mutate(),summarise(), sowie alle mathematischen Funktionen haben Vektoren als Argumente, so dass es sinnvoll ist diese Struktur, zu erhalten.Außerdem kann der Datentyp (logical, integer, double, character, date, factor, etc) von R auch nur dann richtig erkannt werden, wenn die Merkmale richtig definiert sind, also einem Merkmal genau eine Spalte zukommt.
Die Pakete
dplyr,ggplot2und die meisten anderen Pakete des Tidyverse sind darauf ausgelegt, mit bereinigten Daten umzugehen.
Beispiel
Zwei Beispiele, wie elegant interessante Kenngrößen aus bereinigten Daten berechnet werden können.
# Erzeuge die Rate pro 10.000:
table1 |> mutate(rate = cases/population * 10000)# A tibble: 6 × 5
country year cases population rate
<chr> <dbl> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071 0.373
2 Afghanistan 2000 2666 20595360 1.29
3 Brazil 1999 37737 172006362 2.19
4 Brazil 2000 80488 174504898 4.61
5 China 1999 212258 1272915272 1.67
6 China 2000 213766 1280428583 1.67 # Fälle pro Jahr:
table1 |> count(year, wt = cases) # A tibble: 2 × 2
year n
<dbl> <dbl>
1 1999 250740
2 2000 2969209.3 Pivotieren
9.3.1 Die Funktion pivot_longer()
Häufig finden wir in der Praxis Datentabellen vor, bei denen der Spaltenname nicht einem Merkmal entspricht, sondern den Werten eines Merkmals (wie die beiden Tabellen in (4) im Quiz).
-
Mit der Funktion
pivot_longer()kann aus einer solchen Datentabelle eine lange Datentabelle mit mehr Zeilen und zwei neuen Spalten generiert werden.- Die erste Spalte der neuen Datentabelle enthält jeweils die Namen der alten Merkmale als Werte. Das so gewonnene Merkmal, die neue Spalte, kann mit dem Argument
names_to=umbenannt werden. - Die zweite Spalte der neuen Datentabelle enthält jeweils die Werte der alten Merkmale. Diese Spalte kann mit dem Argument
values_to=umbenannt werden.
- Die erste Spalte der neuen Datentabelle enthält jeweils die Namen der alten Merkmale als Werte. Das so gewonnene Merkmal, die neue Spalte, kann mit dem Argument
-
Notwendige Argumente der Funktion
pivot_longer()sind-
data=, die Datentabelle und -
cols=, die Merkmale, die pivotiert werden sollen. Dabei gibt es verschiedene Möglichkeiten bzw. Hilfsfunktionen wie diese angegeben werden können.
-
# Ausführliche Beschreibung und Beispiele:
vignette("pivot")Neben dem bereinigen ungeeigneter Tabellen ist es auch manchmal notwendig lange Tabelle für das Erstellen von bestimmten Graphiktypen zu erzeugen.
Beispiel
Gegeben ist folgende Tabelle
population# A tibble: 3 × 4
country `1960` `1980` `2000`
<fct> <dbl> <dbl> <dbl>
1 France 45865699 54053224 59387183
2 Germany 73179665 78159527 81895925
3 Italy 49714962 56336446 57147081Offenbar ist diese nicht bereinigt im obigen Sinn, da die Jahreszahlen selbst Ausprägungen eines Merkmals sind und keine Merkmale. Mit Hilfe der Funktioon pivot_longer() kann dies nun wie folgt geändert werden
population_long <- population |>
pivot_longer(cols = 2:4,
names_to = "year",
values_to = "population")
population_long # A tibble: 9 × 3
country year population
<fct> <chr> <dbl>
1 France 1960 45865699
2 France 1980 54053224
3 France 2000 59387183
4 Germany 1960 73179665
5 Germany 1980 78159527
6 Germany 2000 81895925
7 Italy 1960 49714962
8 Italy 1980 56336446
9 Italy 2000 57147081population_long <- population |>
pivot_longer(cols = 2:4,
names_to = "year",
values_to = "population",
names_transform = list(year= as.integer))
population_long # A tibble: 9 × 3
country year population
<fct> <int> <dbl>
1 France 1960 45865699
2 France 1980 54053224
3 France 2000 59387183
4 Germany 1960 73179665
5 Germany 1980 78159527
6 Germany 2000 81895925
7 Italy 1960 49714962
8 Italy 1980 56336446
9 Italy 2000 57147081Das gleiche Ergebnis erhält man mittels
population_long <- population |>
pivot_longer(cols = 2:4,
names_to = "year",
values_to = "population") |>
mutate(across(year, as.integer))pivot_longer(): das Argument cols=
Ganz analog zu select() oder der Funktion across() gebt es auch bei den Funktionen pivot_longer() die Spalten/Merkmale für das Argument cols= mit Hilfe der tidyselect-Funktionen auszuwählen.
- Spaltennummer als Vektor (ganzzahlig)
- Spaltennamen als Vektor (Zeichenketten)
- Angabe Spaltennamen in der Form
VON:BIS(ohne Anführungszeichen, es sei denn es sind keine Standard-R Namen, dann Backticks). - die Funktionen aus dem
tidyselect-Paket, von denen einige sehr praktische bereits im Kapitel Kapitel 8.5 erklärt wurden:-
starts_with(), -
ends_with(), -
num_range(),
-
contains(), where()- durch logischen Ausdruck, z.B. Ausschluss von Merkmalen
-
Bei all den Auswahlmöglichkeiten ist immer darauf zu achten, welche Argumente erwartet werden: Zeichenketten oder die direkte Merkmalsbezeichnung ohne Anführungszeichen! Die folgenden Beispiele liefern alle das gleiche Ergebnisse. Die Argumente names_to= und values_to= werden der Übersicht halber weggelassen.
Alle Spaltennamen in einem Vektor
population |>
pivot_longer(cols = c("1960","1980","2000"))Spalten von 1960 bis 2000
Es müssen Backticks benutzt werden, da Spaltennamen nicht Standard R-Namen sind (wir erinnern uns, R-Namen dürfen nicht mit Zahlen beginnen!)
population |>
pivot_longer(cols = `1960`:`2000`)Spaltennamen endet mit 0:
population |>
pivot_longer(cols = ends_with("0"))Alle Spalten, die numerisch sind
Hierbei ist ein wenig Vorsicht geboten, da nicht der Name des numerisch Merkmals ist, sondern das Merkmal selbst!
population |>
pivot_longer(cols = where(is.numeric))Alle Spalten außer country:
population |>
pivot_longer(cols = !country)9.3.2 Grafiken für bereinigte Datentabellen
Bemerkungen:
Lange Datentabellen sind oft besser geeignet, um Daten verschiedene Merkmale gleichzeitig in einer Grafik darzustellen. Wenn z.B. der Name von Aktien statt in Spalten (für jede AG eine Spalte) als Wert in einer neuen Spalte steht, kann diese Spalte einer Ästhetik, wie z.B. der Farbe, zugeordnet werden.
Achtung: Im aktuellen Beispiel (
populationnachpopulation_longwerden numerische Spaltennamen (1960, 1980 und 2000) zu Ausprägungen des MerkmalsJahr. Das MerkmalJahr¸ ist danach allerdings eine Zeichenkette, da es die Spaltennamen waren, und nicht numerisch (was man vielleicht erwartet). Mitmutate()und einer Funktion der Funktionsklasseas.*kann ein Datentyp umgewandelt werden.
Beispiel:
Rechts ist die zuvor erzeugte Tabelle graphisch dargestellt. Im ursprünglichen Datensatz population waren 1960, 1980 und 2000 jeweils Merkmale mit den jeweiligen drei Werten. Nach Verwendung von pivot_longer() können die Daten zusammen in der gewünschten Form dargestellt werden.
population_long |> mutate(year = as.numeric(year)) |>
ggplot(aes(x = year, y = population, colour = country)) +
geom_line() +
geom_point()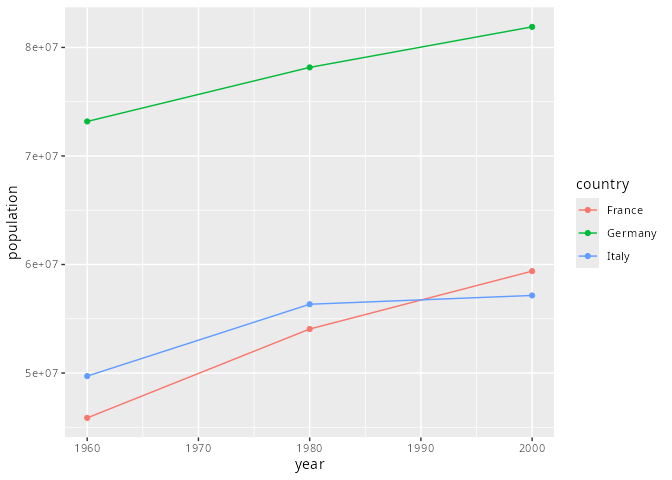
9.3.3 Die Funktion pivot_wider()
Die Funktion pivot_wider() ist das Gegenstück zu pivot_longer() und fasst mehrere Zeilen zu einer zusammen, was die Tabelle breiter (und kürzer) macht. Wir verwenden sie, wenn eine Beobachtung über mehrere Zeilen verteilt ist (wie bei (2) im Quiz).
vignette("pivot")Die wichtigsten Argumente von pivot_wider() sind: * data= Datentabelle, * names_from= Spalte(n) aus data, aus deren Werten die Namen der neuen Merkmale in der breiten Datentabelle gebildet werden sollen. * values_from= gibt an, woher die Werte der neuen Merkmale (Spalten) kommen. * values_fill= (optional), ein Wert, der angibt, wie fehlende Werte aufgefüllt werden.
population_long# A tibble: 9 × 3
country year population
<fct> <int> <dbl>
1 France 1960 45865699
2 France 1980 54053224
3 France 2000 59387183
4 Germany 1960 73179665
5 Germany 1980 78159527
6 Germany 2000 81895925
7 Italy 1960 49714962
8 Italy 1980 56336446
9 Italy 2000 57147081population_long |>
pivot_wider(names_from = year,
values_from = population)# A tibble: 3 × 4
country `1960` `1980` `2000`
<fct> <dbl> <dbl> <dbl>
1 France 45865699 54053224 59387183
2 Germany 73179665 78159527 81895925
3 Italy 49714962 56336446 571470819.4 Trennen von Merkmalen
9.4.1 Die Funktionen separate_wider_*()
In nicht bereinigten Daten kommt es vor, dass in einer Spalte mehrere Merkmale zusammen dargestellt werden (siehe z.B. (3) im Quiz), und manchmal ist es auch sinnvoll bei bereits bereinigten Daten aus einem Merkmal mehrere zu machen: so kann es beispielsweise sinnvoll sein ein Datum in Tag, Monat und Jahr zu trennen oder eine eine Adresse in straße und Hausnummer zu zerlegen.
Es gibt drei verschiedene Funktionen um Daten zu trennen.
separate_wider_delim()
Am einfachsten ist es sich die Funktion an einigen Beispielen zu veranschaulichen. Dfür fangen wir mit einem einfachen, aber oft auftretenen Fall an.
df1 <- tribble(~team, ~ergebnis,
"C" , "1:3:4",
"D" , "4:2:2",
"E" , "3:3:2")
df1# A tibble: 3 × 2
team ergebnis
<chr> <chr>
1 C 1:3:4
2 D 4:2:2
3 E 3:3:2 In der obigen Datenbtabelle ist das Merkmal ergebnis als Zeichenkette in einem ungünstigen Format angegeben, nämlich in der Form Gewonnen:Unentschieden:Verloren, wobei der Trennen offenbar der Doppelpunkt : ist. Besser ist es drei Merkmale G, U und V zu haben, was man mit der Funktion separate_wider_delim() wie folgt erreichen kann.
df1 |> separate_wider_delim(ergebnis,
delim = ":",
names = c("G", "U", "V"))# A tibble: 3 × 4
team G U V
<chr> <chr> <chr> <chr>
1 C 1 3 4
2 D 4 2 2
3 E 3 3 2 Die folgenden Argumente sind notwendig:
-
cols=: das Merkmal (oder auch mehrere Merkmale), diese können mit Hilfe der tidyselect-Funktionen ausgewählt werden, -
delim=: der Trenner, -
names=: die neuen Merkmalsnamen.
Alternativ zu names= kann auch names_sep= verwendet werden. Dies ergibt dann Sinn, wenn mehrere Merkmale gleichzeitig ausgewählt wurden oder eine ungewisse Anzahl an neuen Merkmalen erstellt werden muss. Dabei werden die neuen Merkmalsnamen automatisch vergeben.
separate_wider_position()
Eine zweite Möglichkeit die Merkmale zu trennen ist die Funktion
bei der nicht der Trennen angegeben wird, sondern die Längen der Ausprägungen. Das obige Beispiel kann über den folgenden Ausdruck getrennt werden
df1 |> separate_wider_position(ergebnis,
width = c(G = 1, 1,
U = 1, 1,
V = 1))# A tibble: 3 × 4
team G U V
<chr> <chr> <chr> <chr>
1 C 1 3 4
2 D 4 2 2
3 E 3 3 2 Die Einträge im Argument width= geben die Breite des jeweiligen Merkmals an. Diese benötigen zwingend einen neuen Merkmalsnamen (hier: G, U und V). Die zweite und vierte Zahl in dem Vektor ist die Breite des jeweiligen Trenners.
ein weiteres Beispiel
df2 <- tribble(~Zeit, ~Name,
"26,27:85", "Paul Tergat",
"26-22-75", "Haile Gebrselassie",
"26m20s31", "Kenenisa Bekele",
"26:17,54", "Kenenisa Bekele",
"26:11,00", "Joshua Cheptegei"
)
df2 |> separate_wider_position(Zeit, widths = c(M = 2, 1,
S = 2, 1,
HS = 2))# A tibble: 5 × 4
M S HS Name
<chr> <chr> <chr> <chr>
1 26 27 85 Paul Tergat
2 26 22 75 Haile Gebrselassie
3 26 20 31 Kenenisa Bekele
4 26 17 54 Kenenisa Bekele
5 26 11 00 Joshua Cheptegei Wie man an dem letzten Beispiel sieht besteht ein Vorteil darin, dass es egal ist wie der Trenner aussieht. Die Methode funktioniert auch dann, wenn dieser nicht einheitlich ist.
separate_wider_regex()
Eine letzte Variante um Mermale zu trennen ist mit Hilfe von regulären Ausdrücken. Die Syntax der Funktion davon lautet
separate_wider_regex(
data,
cols,
patterns,
...,
names_sep = NULL,
names_repair = "check_unique",
too_few = c("error", "debug", "align_start"),
cols_remove = TRUE
)Die Verwendung ist sehr ähnlich zu separate_wider_position(), wobei nicht die Länge des jeweiligen Ausdrucks angegeben wird, sondern ein regulärer Ausdruck, der diesen repräsentiert.
Um das erste Beispiel damit zu behandeln könnten wir folgende Funktion verwenden
df1 |> separate_wider_regex(ergebnis, c(G = ".", ".",
U = ".", ".",
V = "."))# A tibble: 3 × 4
team G U V
<chr> <chr> <chr> <chr>
1 C 1 3 4
2 D 4 2 2
3 E 3 3 2 Dies ist in sofern sehr einfach als dass alle Ergebnisse von der gleichen Länge sind, und wir durch den . nur ein Zeichen verwenden, es uns sogar egal sein kann von welchem Typ dieser ist.
Interessanter ist bereits das Beispiel aus der obigen Übung. Dies können wir schreiben als
Orte |> separate_wider_regex(PLZOrt,
patterns = c(PLZ = "\\d+", "[:blank:]",
Ort = "[:alpha:]+"))# A tibble: 5 × 2
PLZ Ort
<chr> <chr>
1 86199 Augsburg
2 52076 Aachen
3 20095 Hamburg
4 86856 Schwabmünchen
5 24111 Kiel
9.4.2 Die Funktionen separate_longer_*()
Neben den Funktionen separate_wider_*() gibt es auch die Funktionen separate_longer_delim() und separate_longer_position(). Dies ist genau dann sinnvoll, wenn die Einträge in einem Merkmal getrennt werden müssen, sie aber einem Merkmal zuzuordnen sind. Bei unserem vorherigen Beispiel mit den Postleitzahlen und den Orten war das zum Beispiel nicht so, da diese verschiedenen Merkmalen PLZ und Ort zugewiesen wurden.
Beispiel
Wir wollen uns das wieder an einem Beispiel klar machen
konzerte <- tribble(~Band, ~Orte,
"Brutalismus 3000", "Berlin, Hamburg, München",
"Edwin Rosen", "Hamburg, Köln, Dortmund, Essen",
"Grande Amore", "Barcelona, Madrid")
konzerte# A tibble: 3 × 2
Band Orte
<chr> <chr>
1 Brutalismus 3000 Berlin, Hamburg, München
2 Edwin Rosen Hamburg, Köln, Dortmund, Essen
3 Grande Amore Barcelona, Madrid Was man an dieser Stelle haben möchte ist eine Datentabelle in der es das Merkmal Band und das Merkmal Ort gibt.
konzerte |> separate_longer_delim(Orte, delim = ", ")# A tibble: 9 × 2
Band Orte
<chr> <chr>
1 Brutalismus 3000 Berlin
2 Brutalismus 3000 Hamburg
3 Brutalismus 3000 München
4 Edwin Rosen Hamburg
5 Edwin Rosen Köln
6 Edwin Rosen Dortmund
7 Edwin Rosen Essen
8 Grande Amore Barcelona
9 Grande Amore Madrid Da keine neuen Merkmale erzeugt werden müssen, wird der alte Merkmalsname übernommen und wir müssen, falls notwendig, mit rename() diesen umbenennen.
Für geeignete Daten existiert auch die Funktion separate_longer_position(), die bei einer festen Position schneidet.
df <- tibble(id = 1:3, x = c("ab", "def", ""))
df# A tibble: 3 × 2
id x
<int> <chr>
1 1 "ab"
2 2 "def"
3 3 "" df |> separate_longer_position(x, 1)# A tibble: 5 × 2
id x
<int> <chr>
1 1 a
2 1 b
3 2 d
4 2 e
5 2 f df |> separate_longer_position(x, 2)# A tibble: 3 × 2
id x
<int> <chr>
1 1 ab
2 2 de
3 2 f df |> separate_longer_position(x, 2, keep_empty = TRUE)# A tibble: 4 × 2
id x
<int> <chr>
1 1 ab
2 2 de
3 2 f
4 3 <NA>
9.5 Vereinen von Merkmalen unite()
Das Gegenstück zu den separate_wider_*() Funktionen ist die Funktion unite(), mit der man Spalten (=Merkmale) zusammenfügen kann.
- Die wichtigsten Argumente von
unite()lauten:-
data=ist eine Datentabelle. -
col=(Zeichenkette) ist der Name der neuen Spalte. -
...Aufzählung der Spalten, die vereint werden sollen. -
sep=ist der Separator, der zwischen den Werten in der neuen Spalte verwendet werden soll. Standard istsep = "_".
- Mit
remove=kann eingestellt werden, ob die alten Spalten aus der Datentabelle entfernt werden sollen. Standard ist hierremove = TRUE.
-
df <- tribble(~Jahrhundert, ~Jahr, ~name,
19, 56, "Ian",
19, 59, "Andrew",
17, 70, "Ludwig")
df |> unite("jahr",
Jahrhundert, Jahr,
sep = "")# A tibble: 3 × 2
jahr name
<chr> <chr>
1 1956 Ian
2 1959 Andrew
3 1770 Ludwig
9.6 Bemerkung zu ... bei Funktionsargumenten
Das ...-Argument ist ein sehr praktisches Argument bei R-Funktionen. Es erlaubt eine unbestimmte Anzahl von Argumenten mit unbestimmten Namen an eine Funktion zu übergeben.
Im obigen Beispiel können zum Beispiel alle Spaltennamen separat hintereinander übergeben werden, aber auch eine Übergabe der Spaltennamen als Vektor mit c() ist erlaubt.
Die Nummer (1) aus der Frage am Anfang des Kapitels wurde mit Hilfe des folgenden Codes erstellt.
table1 <- tribble(
~country, ~year, ~cases, ~population,
"Afghanistan", 1999, 745, 19987071,
"Afghanistan", 2000, 2666, 20595360,
"Brazil", 1999, 37737, 172006362,
"Brazil", 2000, 80488, 174504898,
"China", 1999, 212258, 1272915272,
"China", 2000, 213766, 1280428583
)9.7 Fehlende Werte
- Es gibt zwei Arten von fehlenden Werten: - explizit fehlende Werte, bei denen ein `NA} in der Datentabelle steht - implizit fehlende Werte: Daten, die nicht im Datensatz vorkommen.
Beispiel
Im folgenden Beispiel fehlen zwei Werte:
bestand <- tibble(
jahr = c(2020, 2020, 2020, 2020, 2021, 2021, 2021),
quartal = c( 1, 2, 3, 4, 2, 3, 4),
wert = c(1.88, 0.59, 0.35, NA, 0.92, 0.17, 2.66)
)
bestand# A tibble: 7 × 3
jahr quartal wert
<dbl> <dbl> <dbl>
1 2020 1 1.88
2 2020 2 0.59
3 2020 3 0.35
4 2020 4 NA
5 2021 2 0.92
6 2021 3 0.17
7 2021 4 2.66- ein expliziter:
NAim 4. Quartal 2020, - ein impliziter: das 1. Quartal 2021.
Es hängt nun von der Anwendung ab, ob man eine explizit fehlende Werte löschen möchte (und sie damit evtl. zu implizit fehlenden Werten machen möchte) oder ob man implizit fehlende Werte ergänzen möchte (und sie damit evtl. zu explizit fehlenden Werten macht).
# In dieser Darstellung sieht man beide NAs.
bestand |> pivot_wider(names_from = jahr,
values_from = wert)# A tibble: 4 × 3
quartal `2020` `2021`
<dbl> <dbl> <dbl>
1 1 1.88 NA
2 2 0.59 0.92
3 3 0.35 0.17
4 4 NA 2.66# falls NAs für weitere Betrachtung nicht wichtig ist
# löscht drop_na() die komplette Beobachtung / Zeile
bestand |> drop_na(wert)# A tibble: 6 × 3
jahr quartal wert
<dbl> <dbl> <dbl>
1 2020 1 1.88
2 2020 2 0.59
3 2020 3 0.35
4 2021 2 0.92
5 2021 3 0.17
6 2021 4 2.66
9.7.1 complete()
- Die Funktion
complete()ermöglicht ein Vervollständigen der Datentabelle mit den implizit fehlendenNAs, ohne den Umweg überpivot_wider()pivot_longer()zu gehen. -
complete()erstellt Beobachtungen für jede Kombination der als Argumente angegebenen Merkmale.
bestand <- tibble(
jahr = c(2020, 2020, 2020, 2020, 2021, 2021, 2021),
quartal = c( 1, 2, 3, 4, 2, 3, 4),
wert = c(1.88, 0.59, 0.35, NA, 0.92, 0.17, 2.66)
)
bestand |> complete(jahr, quartal)# A tibble: 8 × 3
jahr quartal wert
<dbl> <dbl> <dbl>
1 2020 1 1.88
2 2020 2 0.59
3 2020 3 0.35
4 2020 4 NA
5 2021 1 NA
6 2021 2 0.92
7 2021 3 0.17
8 2021 4 2.66
9.7.2 fill()
Mit der Funktion fill() werden bei einem Merkmal die expliziten NAs mit dem letzten nicht-NA Wert aufgefüllt.
treatment <- tribble(
~ person, ~ treatment, ~response,
"Vincent Vega", 1, 7,
NA, 2, 10,
NA, 3, 9,
"Mia Wallace", 1, 4,
NA, 2, 9,
NA, 3, 6
)
treatment |> fill(person)# A tibble: 6 × 3
person treatment response
<chr> <dbl> <dbl>
1 Vincent Vega 1 7
2 Vincent Vega 2 10
3 Vincent Vega 3 9
4 Mia Wallace 1 4
5 Mia Wallace 2 9
6 Mia Wallace 3 6