# kann man nicht ausführen, da die Datei nicht existiert...
read_csv2("Beispieldatei.csv", locale = locale(encoding = "WINDOWS-1252"))7 Datenimport
Ein wichtiger Aspekt bei der Analyse von Daten ist der Import der Daten in RStudio. Alle bisher benutzten Datentabellen waren schon Teil der installierten Software. In der Praxis müssen die Daten importiert und bereinigt werden. Alle bisher verwendeten Datentabellen waren (um das Einlesen zu vermeiden) Teil von Paketen. Dies ist im wahren Leben natürlich nicht so: normalerweise geschieht der Datenimports durch das Einlesen von Dateien.
In diesem Kapitel wollen wir genau diesen Aspekt der Datenanalyse behandeln. Andere Methoden des Datenimports, wie z.B. der Import aus einer Datenbank oder aus dem Internet, werden nicht behandelt. Die Konzepte sind aber auch bei diesen Importvarianten die gleichen wie beim Import aus Dateien.
Tidyverse enthält das Paket
readr, mit dem Datentabellen aus Textdateien eingelesen und in Textdateien geschrieben werden können.Das Paket
readxlist geeignet, um Excel-Dateien im xls- oder xlsx-Format einzulesen. Es wird mit demtidyverse-Paket installiert, muss aber zusätzlich geladen werden.Für das Einlesen von SPSS-, Stata- oder SAS-Dateien existiert das Paket
haven, das ebenfalls separat geladen werden muss.
- Für das Lesen und Schreiben von Libre Office / Open Office Dateien im
ODS-Format gibt es das PaketreadODS. Dies ist nicht Teil des Tidyverses und muss extra installiert und geladen werden.
7.1 Einlesen mit readr (csv)
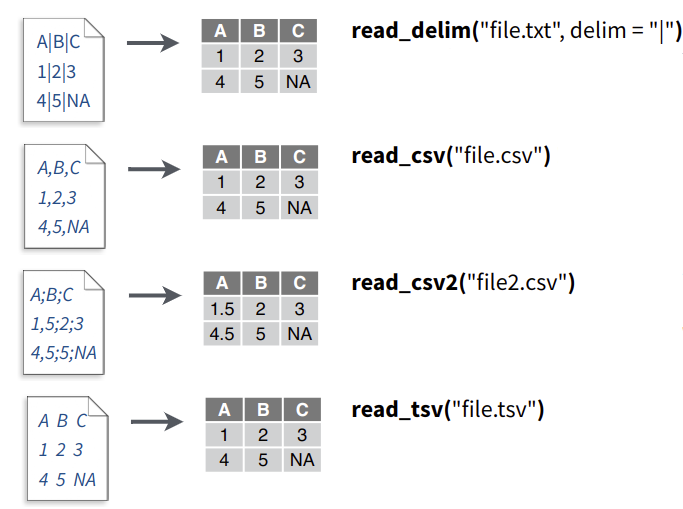
Ein sehr häufiger Dateityp für Daten stellt .csv dar, was für steht comma-separated values steht. Im Gegensatz zu Excel-, Word- oder sonstigen Dateien in einem proprietären Format sind csv-Dateien reine Textdateien und können mit jedem Texteditor geöffnet und gelesen werden. Wie der Namen csv suggeriert, sind die einzelnen Daten mit einem Komma oder einem anderen Trennzeichen getrennt. Im deutschen Sprachraum verwendet man für die Trennung der Merkmale in der REgel das Semikolon ;, da das Komma als Dezimaltrenner bei Fließkommazahlen gebötigt wird. Die unten beschriebenen Funktionen read_csv() bzw. read_csv2() sind aus dem Paket readr. Bei der Auswahl in der Konsole werden auch die Funktionen read.csv() und read.csv2() aus dem Basispaketen vorgeschlagen, jedoch verhalten sich die readr-Funktionen ein wenig anders und sind bei großen Datensätzen auch schneller.
Die Funktion
read_delim()lässt einen beliebigen Spaltentrenner zu. Falls kein Spaltentrenner angegeben wurde, versucht die Funktion, den Spaltentrenner aus den Daten zu ermitteln.read_csv(),read_csv2()undread_tsv()sind Funktionen um Daten einzulesen, bei denen die Spalten mit Kommata, Strichpunkten bzw. Tabulatoren (Taste links neben Q) getrennt sind.
7.1.1 Kodierungen
Sowohl beim Einlesen als auch beim Schreiben von Dateien ist auf die Zeichenkodierung (engl. character encoding oder kurz Encoding) der Daten zu achten. Das Encoding stellt die eindeutige Zuordnung von Zeichen zu Zahlenwerten, die im Computer gespeichert werden, dar. Die Wahl einer falschen Kodierung führt zu falschen Zeichen im Text.
- ASCII (7-Bit-Zeichenkodierung von 1963; Grundlage für spätere Kodierungen),
- ISO 8859-1 / Latin-1 (8-bit Zeichenkodierung, die ASCII um westeuropäische Zeichen erweitert),
- ISO 8859-2 / Latin-2 (8-bit Zeichenkodierung, die ASCII um slawische Zeichen erweitert),
- Windows-1252 / Westeuropäisch / ANSI ist eine 8-Bit Zeichenkodierung für Windows. Sie ist ähnlich zu ISO 8859-1, weicht aber an einigen Stellen ab und
- UTF-8 (Multi-Byte-Zeichenkodierung, die ASCII erweitert und in der Lage ist, alle Unicode-Zeichen, also z.B.kyrillisch und chinesischen Zeichen zu kodieren; de facto Standard für Internetseiten) und viele mehr.
Es gibt mehrere Methoden, beim Einlesen die Kodierung zu berücksichtigen:
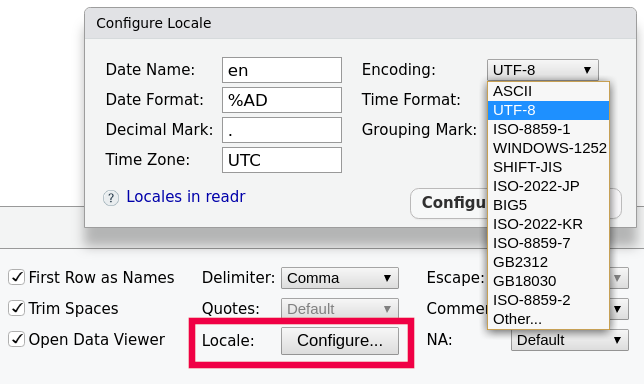
Im Vorfeld können die Dateien mit einem Editor in eine UTF-8-kodierte Datei umkodiert werden. Dann kann (und sollte) man in R prinzipiell immer mit der UTF-8-Kodierung arbeiten. Unter Windows kann man dafür den kostenlosen Editor Notepad++ verwenden.
Liest man die Daten mit Hilfe der
readr-Funktionen ein, so kann die Kodierung überlocaleeingegeben werden. Das Einlesen einer Windows-1252 kodierten, durch Semikolon getrennte csv-Datei sähe dann zum Beispiel so aus:
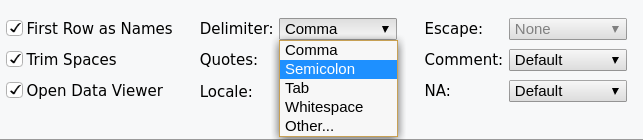
- Nutzt man zum Einlesen der Datei Import Dataset im RStudio, so kann sowohl die Kodierung als auch Spalten- oder Dezimaltrenner etc. über Auswahlmenüs eingestellt werden (siehe Graphiken). Der große Vorteil hierbei ist die Vorschau, mit deren Hilfe man Fehler vermeiden kann.
7.1.2 Fehlende Merkmal- / Spaltennamen und NAs
Für gewöhnlich haben Merkmale Namen und diese sollten in der ersten Zeile einer Datentabelle stehen. Wenn wir mit einer Datentabelle konfrontiert werden, bei denen die Merkmale keine Namen haben, so gibt es auch hier wieder mehrere Möglichkeiten damit umzugehen.
- Liest man die Datei mit einer der
readr-Funktionen ein, so können während des Einlesens mit Hilfe des Argumentscol_names=Merkmalsnamen vergeben werden.
# hier: 3 Merkmale, nach dem Einlesen heißen die Merkmale A, B und C
df <- read_csv2("DateiOhneKopfzeile.csv", col_names = c("A","B","C")) Dabei ist darauf zu achten, dass die Anzahl der Namen mit den tatsächlichen Anzahl an Merkmalen (Spalten) übereinstimmt.
- Eine weitere Möglichkeit, wie man
col_names=verwenden kann, besteht in:
df <- read_csv2("DateiOhneKopfzeile.csv", col_names = FALSE)Dadurch werden die Spaltennamen X1, X2, X3, usw.vergeben. Diese können dann im Weiteren zum Beispiel mit der Funktion rename() umbenannt werden.
- In csv-Dateien kann man fehlende Werte durch leere Zellen oder/und durch eine bestimmte Zeichenfolge kennzeichnen. Leere Zellen werden immer als fehlende Werte erkannt; alle anderen Zeichenketten können über das Argument
na.strings=angegeben werden.
df <- read_csv2("DateiMit999und-alsNA.csv", na = c("999","-"))
# ersetzt 999 und "-"-Zeichen durch NA
7.2 Einlesen mit readxl (Excel)
Ein sehr gängiges Format sind Excel-Tabellen. Um Daten aus Exceldateien einlesen zu können, muss das readxl-Paket geladen werden. Um die Kodierung muss man sich seit Excel 2010 nicht mehr kümmern, da die Exceldateien UTF-8 kodiert sind.
Um Exceldateien ein erstes Mal zu importieren bietet es sich an dies mit - Die Daten aus Exceldateien importiert man am einfachsten über das RStudio-Auswahlmenü Import Dataset.
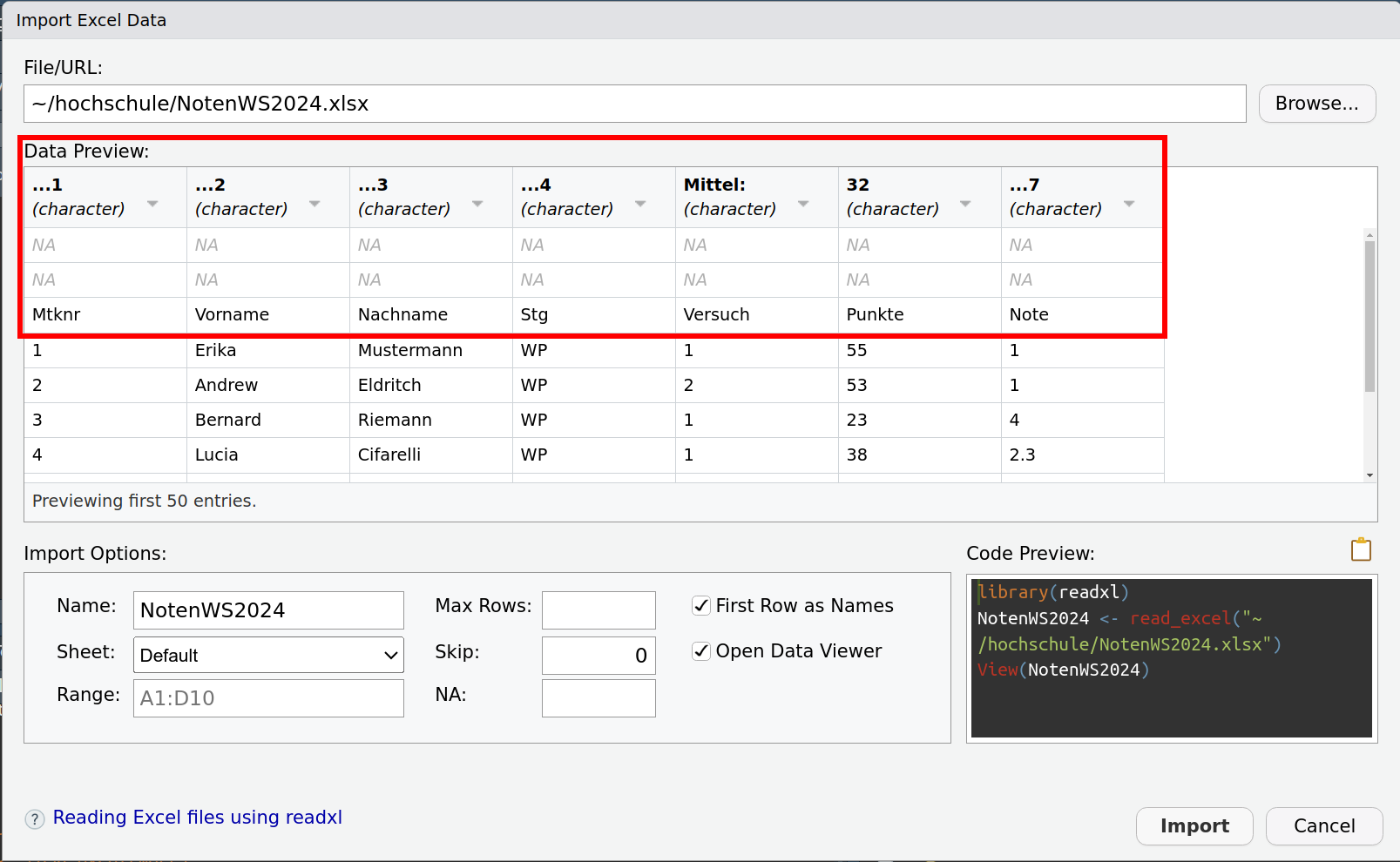
Über eine Oberfläche kann der Import an die jeweilige Excel-Datei angepasst werden. So kann das Arbeitsblatt ausgewählt. Die Abbildung 7.3 zeigt ein Beispiel bei dem die Merkmalsnamen nicht in der obersten Zeile auftauchen.
Wir möchten die Datei aber so einlesen, dass zumindest die Mermalsnamen übernommen werden. Dazu können führende Zeilen ausgelassen werden. In Abbildung 7.4 können wir sehen, dass Das Feld Skip dies möglich macht. Die Vorschau wird nach Eingabe einer Zahl dort gleich aktualisiert. In dem abgebildeten Beispiel müssen wir die ersten drei Zeilen auslassen, damit die Namen der Merkmale richtig erkannt werden.
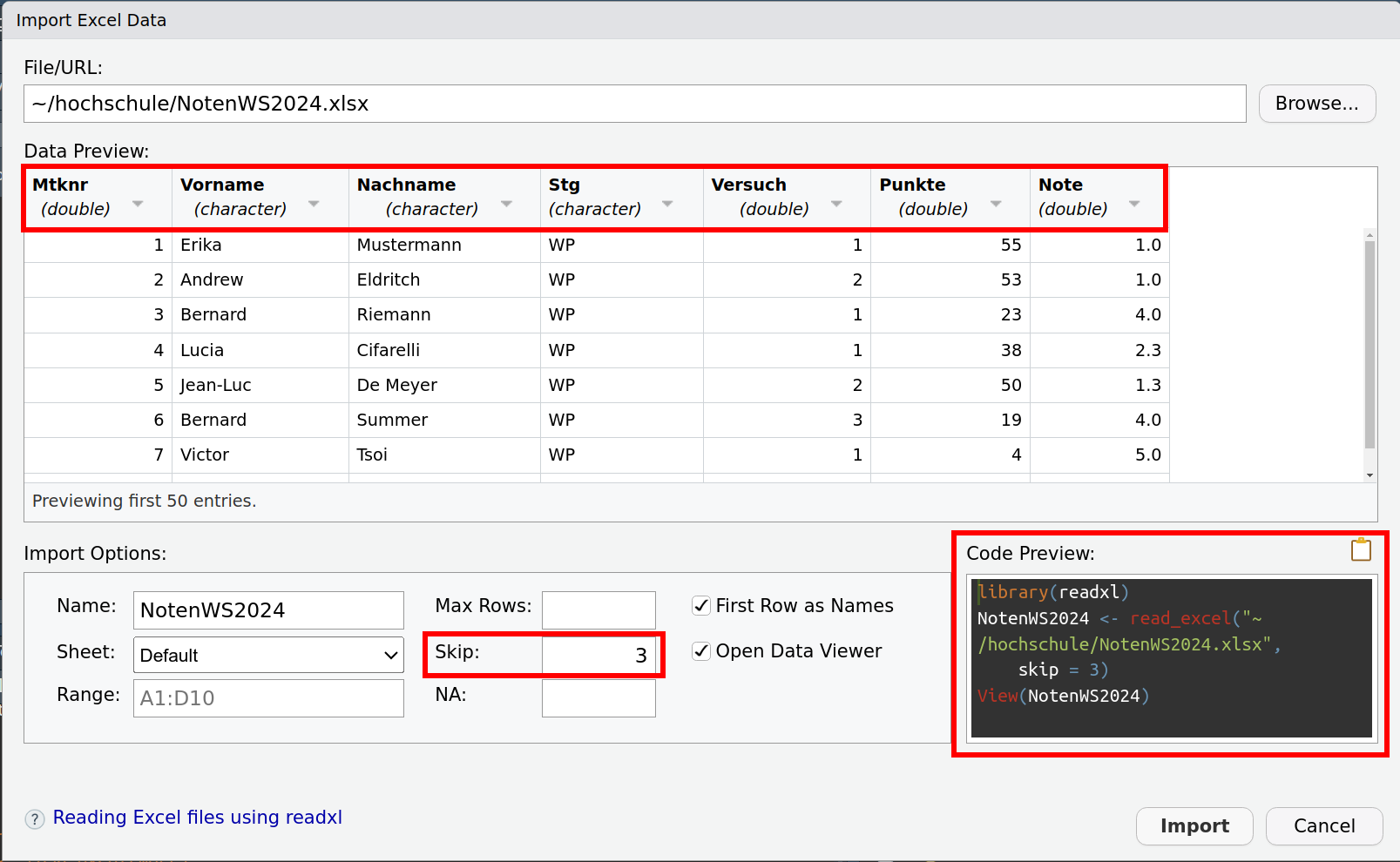
Am Ende kann die Befehlssequenz aus dem Fenster kopiert werden mit der die Excel-Tabelle eingelesen wird. Dies ist dafür gedacht, dass diese in ein Skript geschrieben werden kann, so dass für zukünftige Einlesevorgänge des gleichen Typs das Skript verwendet werden kann.
7.3 Daten parsen
7.3.1 Die parse_*-Funktionen
Die Funktionen
parse_*()wandeln Zeichenketten (<chr>) in spezielle Vektoren wie zum Beispiel logische, numerische (integer oder double) oder Datums- bzw. Zeitangaben um.Im ersten Argument der
parse_*-Funktionen übergibt man einen Vektor mit den umzuwandelnden Zeichenketten, optional kann über das Argumentna=definiert werden, welche Zeichenketten inNAumzuwandeln sind.
Beispiele
logi [1:3] TRUE FALSE NA int [1:4] 1 231 NA 456 Date[1:2], format: "2022-04-01" "1963-03-27" Factor w/ 3 levels "rot","gelb","blau": 1 1 2 1 3parse_factor(c("rot","rot","gelb","rot","blau"))[1] rot rot gelb rot blau
Levels: rot gelb blauBemerkung zur Funktion str()
- Die links benutzte Funktion
str()zeigt die Struktur von beliebigen R-Objekten. Würde man die Funktion nicht nutzen, würde lediglich das Objekt (hier: Vektoren) auf die herkömmliche Weise ausgegeben, wie man beim letzten Beispiel sieht.
- Das Angezeigte entspricht damit im Wesentlichen der Darstellung im Environment des RStudios (‘oben rechts’).
7.3.2 parser-Funktionen
parse_logical()undparse_integer()parsen einen Vektor mit logischen bzw.ganzzahligen Werten.parse_double()undparse_number()sind Parser für Dezimalzahlen. Es ist darauf zu achten, dass der korrekte Dezimaltrenner übergeben wird. Beim Parserparse_number()werden nicht-numerische Zeichen (wie z.B. € vor und nach der ersten Zahl und Gruppierungszeichen (wie z.B. der Tausenderpunkt) ignoriert.parse_character()liest Zeichenketten ein. Dabei muss auf die Kodierung geachtet werden.parse_factor()erstellt Faktoren, das ist die R-Struktur für kategoriale Merkmale mit festen und bekannten Ausprägungen, wie z.B. Zufriedenheitsstufen.parse_datetime(),parse_date()undparse_time()erlaubt es, verschiedene Daten und Zeiten zu parsen. Die vielen Möglichkeiten, wie Daten und Zeiten geschrieben werden, können über das Argumentformat=abgebildet werden.
**Fehler: Entstehen beim Parsen Fehler, so erhält man eine Warnung und die betroffenen Werte werden zu NAs. Bei zu vielen Fehlern hilft die Funktion problems().
x <- parse_integer(c("123", "345", "abc", "123.45"))Warning: 2 parsing failures.
row col expected actual
3 -- no trailing characters abc
4 -- no trailing characters 123.45x[1] 123 345 NA NA
attr(,"problems")
# A tibble: 2 × 4
row col expected actual
<int> <int> <chr> <chr>
1 3 NA no trailing characters abc
2 4 NA no trailing characters 123.45problems(x)# A tibble: 2 × 4
row col expected actual
<int> <int> <chr> <chr>
1 3 NA no trailing characters abc
2 4 NA no trailing characters 123.457.3.3 Parsen von Zahlen
Probleme beim Parsen von Zahlen:
- Dezimaltrenner sind in verschiedenen Ländern und Regionen unterschiedlich.
- Zahlen tauchen oft mit zusätzlichen Zeichen auf, zum Beispiel 100\% oder \$50.
- Zur besseren Lesbarkeit werden bei großen Zahlen auch Gruppentrenner eingefügt, auch diese variieren je nach Region.
An den Beispielen kann man sehen, wie man mit dem jeweiligen Problem umgehen kann.
# Dezimaltrenner:
parse_double("1.23")[1] 1.23parse_double("1,23", locale = locale(decimal_mark = ","))[1] 1.23# Zusätzliche Zeichen:
parse_number("$100")[1] 100parse_number("20%")[1] 20parse_number("It cost $123.45")[1] 123.45# Verschiedene Gruppentrenner:
parse_number("$123,456,789")[1] 123456789parse_number("123.456.789", locale = locale(grouping_mark = "."))[1] 123456789parse_number("123'456'789", locale = locale(grouping_mark = "'"))[1] 1234567897.3.4 Parsen von Zeichenketten
Man könnte denken, Zeichenketten zu parsen sei einfach, da nur die Zeichenkette zurückgegeben werden soll, allerdings spielt hier die Kodierung eine Rolle, was zu einem Problem werden könnte. Die UTF-8 Kodierung löst prinzipiell alle diese Probleme, da es für jedes Zeichen, das von Menschen genutzt wird, eine UTF-8 Kodierung gibt. Das Paket readr geht prinzipiell von einer UTF-8 Kodierung aus.
Was passiert R intern?
Die von R intern verwendete Kodierung ordnet jedem Zeichen eine Hexadezimalzahl zu:
charToRaw("Tarantino")[1] 54 61 72 61 6e 74 69 6e 6fWie erkennt man die Kodierung?
Prinzipiell ist es schwierig, nur aus Daten die Kodierung zu erkennen.
Es gibt aber Hoffnung: manche Programme wie Notepad++ können helfen, oder man nutzt die Funktion
guess_encoding(), die Wahrscheinlichkeiten für Kodierungen angibt.
(x1 <- "El Ni\xf1o was particularly bad this year")[1] "El Ni\xf1o was particularly bad this year"x2 <- "\x82\xb1\x82\xf1\x82\xc9\x82\xbf\x82\xcd"
parse_character(x1, locale = locale(encoding = "Latin1"))[1] "El Niño was particularly bad this year"guess_encoding(charToRaw(x1))# A tibble: 2 × 2
encoding confidence
<chr> <dbl>
1 ISO-8859-1 0.46
2 ISO-8859-9 0.23guess_encoding(charToRaw(x2))# A tibble: 1 × 2
encoding confidence
<chr> <dbl>
1 KOI8-R 0.427.3.5 Parsen von Faktoren
R nutzt Faktoren um kategoriale Merkmale zu speichern. Diese können geordnet sein (ordinale Merkmale) oder ungeordnet (nominale Merkmale).
Mit der Funktion
parse_factor()können kategoriale Merkmale geparsed werden.Das Argument
levels=gibt dabei alle möglichen Ausprägungen vor. Es müssen nicht alle Ausprägungen im zu parsenden Vektor vorkommen, allerdings können nur die inlevels=vorkommenden Ausprägungen geparsed werden (siehe Beispiel), ansonsten wirdNAzurückgeliefert.Das Argument
ordered=TRUEmuss bei geordneten Faktoren gesetzt werden. Die Ordnung ist durch die Reihenfolge im Argumentlevels=vorgegeben (siehe zweites Beispiel).-
Weitere Argumente der Funktion
parse_factor()sind-
na=, bei der man angeben kann, welche Einträge alsNAgewertet werden. Standard istNAund die leere Zeichenkette. -
include_na=gibt an, obNAals eigene Ausprägung gewertet werden soll. Der Standard istTRUE.
-
frucht <- c("Apfel", "Banane", "Mango")
parse_factor(c("Apfel", "Banane", "Zitrone"),
levels = frucht)Warning: 1 parsing failure.
row col expected actual
3 -- value in level set Zitrone[1] Apfel Banane <NA>
attr(,"problems")
# A tibble: 1 × 4
row col expected actual
<int> <int> <chr> <chr>
1 3 NA value in level set Zitrone
Levels: Apfel Banane Mangonoten <- c("gut", "ok", "schlecht")
parse_factor(c("ok", "gut", "ok", "schlecht"),
levels = noten,
ordered = TRUE)[1] ok gut ok schlecht
Levels: gut < ok < schlecht7.3.6 Parsen von Datums- und Zeitangaben
Es gibt drei verschiedene Parser, abhängig davon, was man parsen möchte:
parse_datetime()ist für Datum und Zeit in einer Zelle. Erwartet ist hier ein Eintrag gemäß ISO 8601, dem internationalen Standard. Die Ordnung ist von groß nach klein, also Jahr, Monat, Tag, Stunde, Minute und Sekunde. Zusätzlich kann die Zeitzone angegeben werden.parse_date()für ein Datum. Die erwartete Eingabe ist ein etwas von der FormYYYY-MM-DD, wenn das Argumentformat=nicht angegeben wird.parse_time()für Zeiten. Die erwartete Eingabe ist Stunden,:, Minuten. Optional kann noch:, Sekunden eingegeben werden. Außerdem kannamoderpmangegeben werden, falls keine 24-Stunden-Schema verwendet wird.
Für Zeiten ist es empfehlenswert, das Paket hms zu verwenden, da R base keine gute Klasse für Zeiten hat.
Sollten die Daten nicht in einem geeigneten Format vorliegen (Trenner, Reihenfolge, etc.), so kann bei jeder der obigen Funktionen angegeben werden, im welchem Format Datum oder Zeit vorliegen. Das Argument dazu heißt format= und wird auf der nächsten Seite erläutert.
# T Trenner Datum Zeit, +01 Zeitzone (hier MEZ)
parse_datetime("1969-07-20T2117+01")[1] "1969-07-20 20:17:00 UTC"# ohne Zeitangabe -> Mitternacht
parse_datetime("1994-10-14")[1] "1994-10-14 UTC"# beide Eingaben liefern gleiches Ergebnis:
parse_date("1994-10-14")[1] "1994-10-14"parse_date("1994/10/14")[1] "1994-10-14"
Attache Paket: 'hms'Das folgende Objekt ist maskiert 'package:lubridate':
hmsparse_time("20:00")20:00:00parse_time("01:23 am")01:23:00gaben für parse_datetime(), parse_date(), parse_time()
| Symbol | Bedeutung |
|---|---|
%Y |
Jahr (4-stellig) |
%y |
Jahr (2-stellig) 00-69: 2000 bis 2069, 70-99: 1970 bis 1999 |
%m |
Monat (2-stellig) |
%b |
Monat (abgekürzter Name, z.B. ‘Jan’) |
%B |
Monat (voller Name, z.B. ‘January’) |
%d |
Tag (2-stellig) |
%e |
Tag (optional führende Leerstelle) |
%H |
Stunden 0-23 |
%I |
Stunden 0-12 (muss mit %p verwendet werden) |
%p |
AM / PM |
%M |
Minuten |
%S |
ganzzahlige Sekunden |
%OS |
Sekunden |
%Z |
Zeitzone als Name (z.B. America/Chicago) |
%z |
Offset zu UTC (z.B +0800) |
%. |
lässt eine Nichtziffer aus |
%* |
lässt eine beliebige Anzahl Nichtziffern aus |
Neben dem zu parsenden Vektor kann man das Format mit dem Argument format= angeben. Da es das zweite Argument in den Funktionen parse_*() ist, kann der Argumentenname weggelassen werden.
parse_date("2010-10-01")[1] "2010-10-01"parse_date("01/02/15", format = "%m/%d/%y")[1] "2015-01-02"parse_date("01/02/15", "%d/%m/%y")[1] "2015-02-01"parse_date("01/02/15", "%y/%m/%d")[1] "2001-02-15"parse_date("18.04.22", "%d.%m.%y") |> str() Date[1:1], format: "2022-04-18"Einlesen und parsen
Beim Einlesen einer Datei mit dem Paket readr wird mittels einer Heuristik versucht, den Datentyp jeder Spalte (jedes Merkmals) zu ermitteln. Dies geschieht (intern) mit der Funktion guess_parser(), bei der folgende Regeln hinterlegt sind:
- logisch (logical), falls nur
F,T,FALSEoderTRUEenthalten sind, - ganzzahlig (integer), falls nur numerische Zeichen und as Minuszeichen enthalten sind,
- Fließkommazahl (double), falls nur zulässige Fließkommazahlen (auch exponentialschreibweise, z.B.
4.5e-5) enthalten sind, - Zahl (number), falls zulässige Fließkommazahlen mit Gruppierungen enthalten sind,
- Zeit (time): falls standardisiertes Zeitformat,
- Datum (date): falls standardisiertes Datumformat,
- Datum + Zeit (date-time): jedes zulässige ISO 8601 Format.
In allen anderen Fällen bleibt der Datentyp der Spalte eine Zeichenkette (character).
# Beim Einlesen passiert folgendes:
guess_parser("2010-10-01")[1] "date"guess_parser("15:01")[1] "time"guess_parser(c("TRUE", "FALSE"))[1] "logical"guess_parser(c("1", "5", "9.0"))[1] "double"guess_parser(c("12,352,561"))[1] "number"# parse_guess liefert den geparsten Vektor
str(parse_guess("2010-10-10")) Date[1:1], format: "2010-10-10"7.3.7 Probleme und Lösungen beim Einlesen und Parsen
Diese Vorgehensweise kann zu Problemen führen, gegebenenfalls sind alle NAs sind, so dass die Spalte als logischer Vektor eingestuft wird.
Lösung
- Mit Hilfe der Funktion
problems()kann man die auftretenden Probleme ausfindig machen. - Beim Einlesen können diese Probleme dann bereits behoben werden. Über das Argument
col_types=kann der Datentp explizit vorgegeben werden (Syntax siehe unten). Für die Funktionencol_*()gibt es die gleichen Datentypen wie fürparse_*(). - Tauchen in mehreren Spalten Probleme auf, so ist es ratsam, sich eine Spalte nach der anderen vorzunehmen bis
problems()keine Fehler mehr ausgibt.
Beispiel:
df <- read_csv(readr_example("challenge.csv"),
col_types = cols(
x = col_double(),
y = col_date()
)
)- Man kann auch alle Spalten als Zeichenketten einlesen, etwaige Fehler beheben und die Spalte dann zu einem späteren Zeitpunkt im Skript umwandeln. Dabei hilft neben den
parse_*()-Funktionen auch die Funktiontype_convert().
## alle Spalten als Zeichenkette einlesen:
df3 <- read_csv(readr_example("challenge.csv"),
col_types = cols(.default = col_character()))
type_convert(df3)
── Column specification ────────────────────────────────────────────────────────
cols(
x = col_double(),
y = col_date(format = "")
)# A tibble: 2,000 × 2
x y
<dbl> <date>
1 404 NA
2 4172 NA
3 3004 NA
4 787 NA
5 37 NA
6 2332 NA
7 2489 NA
8 1449 NA
9 3665 NA
10 3863 NA
# ℹ 1,990 more rows7.4 Schreiben einer Datei
Die Funktionen write_csv() und write_tsv()
Das Paket
readrbietet zwei nützliche Funktionen um eine (überarbeitete) Datentabelle wieder zu speichern, nämlichwrite_csv()undwrite_tsv(). Die Vorteile der so abgespeicherte Datentabelle sind,dass die Kodierung UTF-8 ist und
dass Datum bzw. Datum inkl. Zeiten im ISO 8601-Format sind.
Bei einem erneuten Einlesen entstehen damit keine Probleme mehr. * Die wichtigsten Argumente der Funktionen write_csv() und write_tsv() sind
| Argument | Bedeutung |
|---|---|
x |
Datentabelle, die zu speichern ist |
file |
Name der Datei (endet mit .csv / .tsv) |
na |
(optional) wie NAs abgespeichert werden sollen. Standard ist NA. |
write_csv(df3, "df3.csv")
df4 <- read_csv("df3.csv") Rows: 2000 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
dbl (1): x
date (1): y
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.df4 # A tibble: 2,000 × 2
x y
<dbl> <date>
1 404 NA
2 4172 NA
3 3004 NA
4 787 NA
5 37 NA
6 2332 NA
7 2489 NA
8 1449 NA
9 3665 NA
10 3863 NA
# ℹ 1,990 more rows7.4.1 Schreiben von Excel-Dateien
Es gibt mehrere Pakete mit denen man Excel-Dateien schreiben kann kann. Diese müssen zusätzlich instaliert werden:
Das Paket writexl (einfach):
Eine oder mehrere Datentabellen können über den Befehl
write_xlsx()aus dem Paket in eine Excel-Datei geschrieben werden.Für jede Datentabelle wird ein eigenes Arbeitsblatt erzeugt.
Formatierungen, Schriftarten, Rahmen usw. können nicht beeinflusst werden.
Das Paket xlsx (mächtig):
Es benötigt für die Installation eine lauffähigen Java-Installation, das R-Paket
rjava.Mit diesem Paket können auch Formatierungen, Schriftarten, Hintergrundfarben und Rahnmen von Zellen, Zeilen und Spalten gesetzt werden. Damit kann über R ein hübsch formatiertes Excel-Datei, das Daten aus R enthält, erstellt werden.
Details findet man auf den Hilfeseiten, User guides und der Paket-Vignetten.
